Az Árpád-ház filogenetikai eredetének meghatározása III. Béla Y-kromoszóma szekvenciájának elemzése alapján
A cikk angol változata a Nature kiadó European Journal of Human Genetics szakfolyóiratában jelent meg.
Nagy Péter L. 1,2,*, Olasz Judit3, Neparáczki Endre4,5, Nicholas Rouse1,6, Karan Kapuria6, Samantha Cano1,7, Huijie Chen1,6, Julie Di Cristofaro8, Goran Runfeldt9, Natalia Ekomasova10,11, Maróti Zoltán4,12, Jeney János4, Sergey Litvinov10,11, Murat Dzhaubermezov10,11, Lilya Gabidullina10, Szentirmay Zoltán3, Szabados György13,14,15, Dragana Zgonjanin16,17, Jacques Chiaroni8, Doron M. Behar18Elza Khusnutdinova10,11, Peter A. Underhill19, és Kásler Miklós3
1: Korábbi munkahely: Laboratory of Personalized Genomic Medicine, Department of Pathology, Columbia University, New York, New York, USA
2: Jelenlegi munkahely: Praxis Genomics LLC, Atlanta, Georgia, USA
3: Országos Onkológiai Intézet, Budapest, Magyarország
4: Magyarságkutató Intézet, Archeogenetikai Kutatóközpont, Budapest, Magyarország
5: Szegedi Tudományegyetem, Genetikai Tanszék, Szeged, Magyarország
6: MNG Laboratories LLC, Atlanta, Georgia, USA
7: Jelenlegi munkahely: Boston’s Children’s Hospital, Boston, Massachusetts, USA
8: Aix Marseille Université, CNRS, EFS, ADES, "Biologie des Groupes Sanguins", Marseille, France
9: Gene by Gene, Houston, TX, USA
10: Department of Genetics and Fundamental Medicine, Bashkir State University, Ufa
11: Institute of Biochemistry and Genetics – Subdivision of the Ufa Federal Research Centre of Russian Academy of Sciences, Ufa, Russia
12: Szegedi Tudományegyetem, Gyermekgyógyászati Klinika és Gyermekegészségügyi Központ, Szeged, Magyarország
13: Szent István Múzeum, Székesfehérvár, Magyarország
14: Siklósi Gyula Várostörténeti Kutatóközpont, Székesfehérvár, Magyarország
15: Magyarságkutató Intézet, László Gyula Kutatóközpont és Archívum, Budapest, Magyarország
16: Institute of Forensic Medicine, Clinical Center of Vojvodina, Novi Sad, Serbia
17: Faculty of Medicine, University of Novi Sad, Novi Sad, Serbia
18: Estonian Biocentre, Institute of Genomics, University of Tartu, Estonia
19: Department of Genetics, Stanford University, Stanford, CA, USA
A szerzők és társszerzők nyilatkoznak arról, hogy nem áll fenn érdekellentét
Futócím: III. Béla király Y-kromoszóma markereinek elemzése
*Kontaktszemélyként megjelölt szerző:
E-mail:
Absztrakt
Célunk az Árpád-ház eredetének feltárása volt az eredetileg a székesfehérvári székesegyházban eltemetett III. Béla magyar király (1172–11196) és 8 további személy (6 férfi, 2 nő) csontvázmaradványaiból származó DNS-ek genom-szekvenálásával. Az Y-kromoszóma elemzés megállapította, hogy két egyén, III. Béla és HU52 az R-Z2125 haplocsoportba tartozik, amely Dél-Közép-Ázsia térségében gyakori, és másodlagos kiterjedéssel bír a mai Irán, a Volga uráli régió és a Kaukázus terültén. E regiókból származó 4340 egyénből álló mintagyűjteményből 208 olyan egyén genomját szekvenáltuk meg, akik az R-Z2125/R-Z2123-as haplocsoportba tartoztak. Ezekből az adatokból megállapítottuk, hogy az Árpád-ház legközelebbi rokonai a legnagyobb számban az oroszországi Baskíria Burzjan és Abzelil járásaiban élő R-SUR51 haplocsoportba tartozó baskírok. Elemzésünk olyan „egy nukleotidot érintő” (SNP) markereket is feltárt, amelyek egy új, Árpád-ház specifikus (R-ARP) haplocsoportot határoznak meg.
Az R-Z2123 haplocsoport nagy felbontású filogenenetikai feltérképezése alapján az első magyar királyi dinasztia eredete körülbelül 4500 évvel ezelőtt az Észak-Afganisztán környéki régiókban jelölhető meg, és a baskírokat azonosítja legközelebbi rokonként. A két népcsoport szétválásának időpontja az időszámításunk szerinti első évezred elejére tehető.
Kulcsszavak: magyar őstörténet, Árpád-ház, III. Béla király, R1a, R-Z2123, R-SUR51, R-ARP, baskírok
Bevezetés
A magyar államot a Kárpát-medencében megalapító Árpád-ház (kb. 850 – 1301) történelemformáló szerepet töltött be Kelet-Európában. Bár a dinasztiát Álmos herceg (kb. 820 – kb. 894) alapította, mégis a modern történettudományban nevét az ő fiáról, Árpád hercegről (kb. 845 – kb. 907) kapta, aki kb. 894 és kb. 907 között uralkodott és kb. 862 és kb. 896 között vezette be a magyarokat a Kárpát-medencébe. Bár történtek részleges mitokondriális és Y-kromoszóma alapú elemzések a honfoglaló magyar vezetőréteg temetőiben található maradványokból, az Árpád-ház etnikai eredete tudományosan vitatott a hiányzó genetikai bizonyítékok miatt (2-6). Történelmi források alapján 10 Árpád-házi vezetőt, 8 királyt és 2 herceget helyeztek nyugalomra a Szűz Mária-Bazilikában, vagy mai nevén a székesfehérvári koronázóbazilikában az 1543-es török megszállás előtt (7-9). Az ezt követő évszázadok során a háború, és az épület elhanyagolása miatt a bazilika megsemmisült, és csak III. Béla (1148-1196) király és első felesége, Antióchiai Anna (1150–1184/85) királyi sírjai maradtak érintetlenek. A királyi maradványokat 1848-ban fedezte fel a kor vezető régésze és történésze, Érdy János, az MTA tagja és a Budapesti Nemzeti Múzeum kurátora (10). A III. Béla és Antióchiai Anna maradványaival együtt feltárt királyi jelképek a Magyar Nemzeti Múzeum tulajdonában vannak. A maradványokat először 1862-ben a budai Mátyás-templom kriptájában, majd később, 1898-ban a templom Szentháromság-kápolnájában temették újra. 1862 -ben és 1874-ben további maradványokat tártak fel a székesfehérvári bazilika területén, amelyek közül 8-at a Mátyás-templomban helyeztek el 1900-ban. (11)
III. Béla király és 6 másik ismeretlen férfi csontvázának Y-kromoszóma rövid egymásután ismétlődő (STR) marker elemzése arra utalt, hogy a maradványok közül csak egy, a HU52-vel jelölt tartozik az Árpád-házhoz (7).
Az új-generációs szekvenálás (NGS) területén az elmúlt évtizedben bekövetkezett fejlődés alapján lehetővé vált nagyszámú minta mitokondriális és Y-kromoszóma vizsgálatának költséghatékony módon történő elvégzése (12). Két kísérleti kutatás (13, 14) 456, illetve 1244, a világ minden tájáról származó mintát vizsgált (szekvenált) meg és több mint 65 000 Y-kromoszómás SNP-t fedezett fel. A kutatásokról szóló tanulmányok megjelenése óta az ismert Y-kromoszóma SNP-k száma megháromszorozódott, és ez lehetővé tette az apai vonalon meglévő rokoni kapcsolatok magas felbontású elemzését. (15) Elvégeztük az Olasz és mtsai. 2018. által vizsgált leletekből származó DNS NGS-elemzését, es meghatároztuk az Árpád-ház filogenetikai eredetét és legközelebbi rokonságát 40 eurázsiai népesség tagjaival közös haplocsoport markerek alapján.
Anyagok és módszerek
Archaikus minták
Az ebben a tanulmányban vizsgált ősi maradványok a Magyar Katolikus Egyház védelme alatt állnak. Erdő Péter, Esztergom-Budapest érseke írásban járult hozzá, hogy a maradványokon genetikai elemzést végezhessünk, ezt pedig az EMMI 26090/2019/MINKABINET sorszám alatt jegyezte be. A 9 csontvázmaradványból a mintavételt az Országos Onkológiai Intézetben, Budapesten végezték (7). A DNS-izolálása a Johann-Friedrich-Blumenbach Zoológiai és Antropológiai Intézet, Történeti Antropológiai és Humán Ökológiai Tanszékén, a Susanna Hummel laboratóriumban történt (7). Az új-generációs szekvenálás (NGS) és az elsődleges adatelemzés a Praxis Genomics LLC (Atlanta GA) által történt, és az eredményeket a Magyarságkutató Intézet Archeogenetikai Kutatóközpontja megerősítette az összes adat ismételt elemzése révén. Az NGS folyamatról és az adatelemzésről szóló további szakmai információ a „Könyvtár készítés és új-generációs szekvenálás” és az „Adatelemzés” fejezetekben található. A DNS-minták kezelése során a CLIA és CAP irányelveket mindvégig szigorúan betartották.
Jelenkori minták
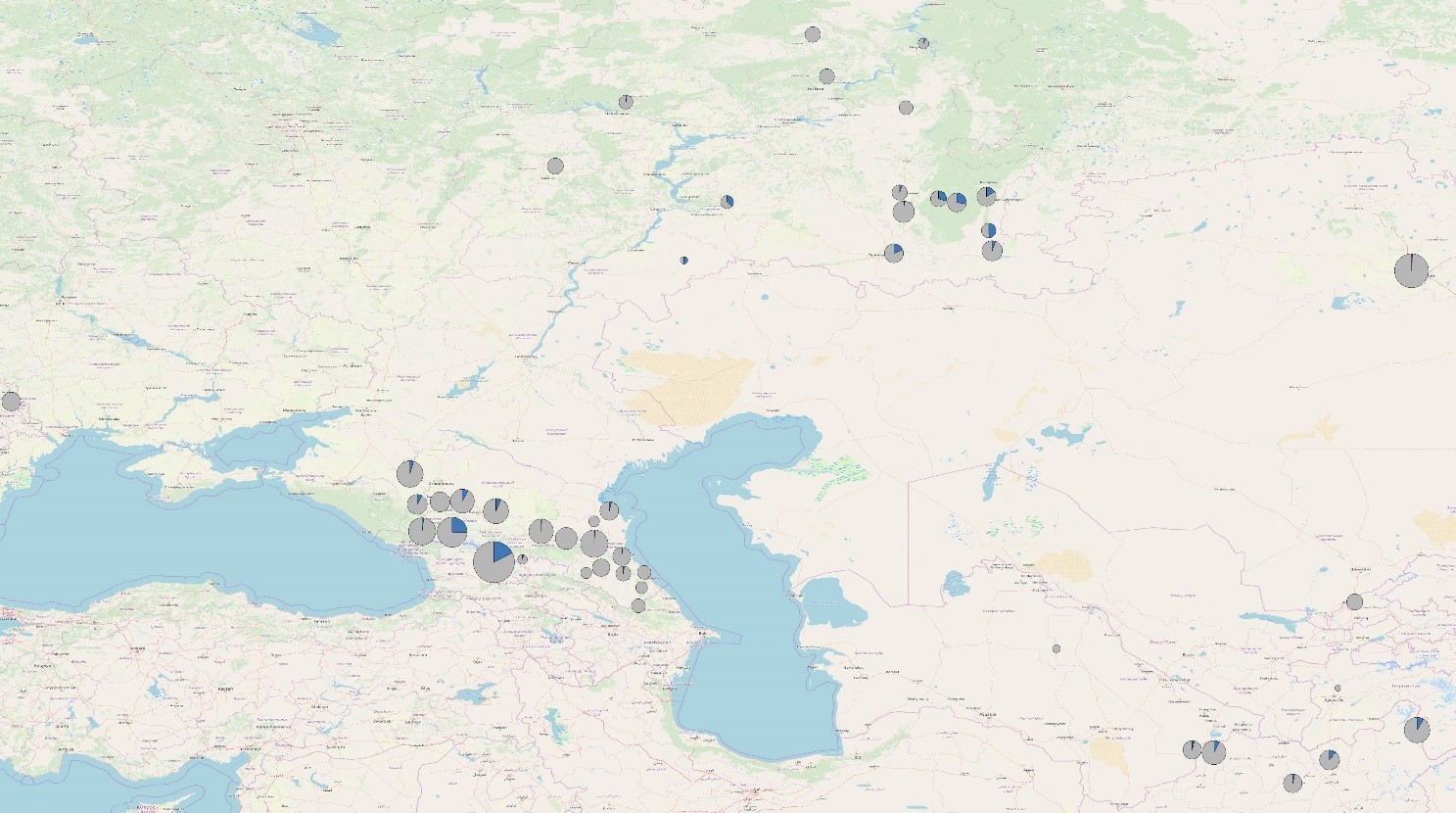
40 különböző népességből származó 4340 jelenkori mintát vizsgáltunk Sanger-féle szekvenálással, hogy azonosítsuk azokat, melyek hordozzák a Z2125 és Z2123 SNP-markereket (1. ábra és S1 táblázat). Az R-Z2123 haplocsoporthoz tartozó, 20 népességből származó 206 reprezentatív minta DNS könyvtárát mindkét végről (paired-end) szekvenáltuk (2x151bp) a Novaseq 6000 (Illumina) gépet használva (1. ábra és S1 táblázat). A minták jelen tanulmányban történő felhasználását az Orosz Tudományos Akadémia UFA Szövetségi Kutatóközpontjának Etikai Bizottsága hagyta jóvá (dokumentumszám: MKI-F/321-1/2019). További technikai információ található a „Könyvtár készítés és új-generációs szekvenálás” fejezetben. Az alanyok kivétel nélkül olyan önkéntes résztvevők voltak, akik írásban járultak hozzá mintáik genetikai elemzés céljára történő felhasználásához.
Két korábban publikált, egy iraki és egy „iraki zsidó” személyhez tartozó Y-kromoszóma szekvenciát is letöltöttünk az Európai Nukleotid Archívumból (http://www.ebi.ac.uk/ena; PRJEB21310 hozzáférési szám) és használtunk fel analízisünkben (16).
Könyvtár készítés és új-generációs szekvenálás
A recens mintákat szonikálással fragmentáltuk a gyártó utasítása szerint (Covaris S220 Ultrasonicator; Woburn, MA) úgy, hogy a fragmentek átlagos hossza kb. 300 bp legyen. Az archaikus DNS mintáknál nem volt szükség szonikálásra. Alacsony molekulasúlyú DNS-t (<300 bp) AMPure XP gyöngyökkel (Beckman Coulter, Indianapolis, IN) dúsítottuk az összes mintából. A könyvtár készítése TruSeq Nano DNA LT kit (Illumina, San Diego, CA) felhasználásával történt a gyártó utasításai szerint. A könyvtárak hosszát és minőségét a Fragment Analyzerrel (Advanced Analytical, Santa Clara, CA), a mennyiségüket qPCR-rel (S1000/CFX96 Real Time System; Biorad, Hercules, CA) ellenőriztük. A könyvtárakat mindkét végröl (paired-end) szekvenáltuk HiSeq 2500 (2x125 bp); és NovaSeq 6000 (2x150bp) (Illumina) szekvenátorokon, a gyártó utasításait követve.
Adatelemzés
Mind az archaikus, mind a jelenkori adatok elsődleges analízise a Praxis Genomics LLC-ben történt. Az új-generációs szekvenálási futások minta-specifikus egységekre való szétválasztása a BaseSpacen történt (17). Adapter és minőségi trimmelés a Trim Galore (Babraham Bioinformatics) programmal történt az összes minta összes fastq fájlján (18). Ezután a 40 bp alatti szekvencia olvasatokat eltávolítottuk, és csak a megfelelő paired-end olvasatokat tartottuk meg és illesztettük GRCh38 referenciagenomhoz a Dragen V.3.2.5. (Illumina) szoftver segítségével a javasolt alapértelmezett beállításokat használva. A variánsok felfedezése is a Dragen szoftverrel történt. A variánsok vizuális ellenőrzése az Integrative Genome Viewer (19, 20) segítségével történt.
Az archaikus genomok Dragen illesztés és variáns azonosítás alapján nyert eredményeit a Magyarságkutató Intézet Archeogenetikai Kutatóközpont analízise is megerősítette. Az archaikus adathalmazt mind a GRCh37 (hs37d5) és a GRCh38 referenciagenomhoz illesztették a Burrow-Wheels-Aligner (v 0.7.17) felhasználásával a MEM paranccsal, úgy, hogy a „reseeding” le volt tiltva. A GRCh37 illesztésnél a PICARD tools-t használták a duplikátumok megjelölésére és csak a megfelelő párokat használtuk a további analíziseknél, amelyek nagyobb mint 90%-os megegyezést mutattak a referencia genommal.
Az archaikus DNS-re jellemző károsodásokat a MapDamage 2.0 (21) programmal becsültük meg. A szekvencia olvasatok minőségi mutatóinak analízisénél a „rescale” opciót használtuk, a poszt-mortem károsodást figyelembe véve. A kromoszómális nem meghatározása Skoglund et al. (22) módszerével történt. A mitokondriális genomkontamináció becslése a Schmutzi algoritmussal történt (23) (1. táblázat). A férfi minták esetében az ANGSD X kromoszóma kontamináció becslés módszerét alkalmaztuk (24).
A mitokondriális haplocsoport meghatározása a HaploGreppel történt (25) (2. táblázat, S2 táblázat). Az Y-kromoszóma haplocsoport meghatározása először a Yleaf-fel történt (26). A genom adatok részletesebb kielemzéséhez a FamilyTreeDNA Y-DNA Haplotree és az ISOGG SNP adatbázisát (27, 28) használtuk a „bam-readcount” program felhasználásával (29). A „bam-readcount” megadja a lefedettséget, a nukleotid összetételt, és a szekvencia olvasatok minőségét a megadott kromoszómális pozíciókban. Ily módon sikerült a Yleaf programnál mélyebb haplocsoport besorolást kapni, és a korábban ismeretlen Árpád-ház (R-ARP) haplocsoport felfedezése is lehetővé vált. A Clustal Omega programot (Version 1.2.4) (30) használtuk az R-Z2123 haplocsoport filogenetikai rekonstrukciójához és a koaleszcens idő kiszámításához a derivált haplocsoportok között. Csak azon SNP-eket használtuk a koaleszcens idő kiszámításához, amelyek egy körülbelül 10 Mb-os régióba esnek az Y-kromoszómán, ahogy Poznik és mtsai. leirták (13). A filogenetikai fát a FigTree programmal (v1.4.4) (31) rajzoltuk meg.
A cikkben vizsgált összes minta mitokondriális es Y-kromoszóma szekvenciája BAM fájl formában elérhető az interneten (https://www.ncbi.nlm.nih.gov/bioproject/PRJNA490697: Determination of the phylogenetic origins of the Árpád Dynasty based on Y chromosome sequencing of Béla the Third.)
Eredmények
Az újgenerációs szekvenálás adatai
9 archaikus DNS minta újgenerációs szekvenálását végeztük el. A mintánkénti egyedi leolvasások száma, az endogén DNS tartalom, az X és Y, illetve az autoszómális kromoszómák lefedettsége igen különböző volt a különböző mintákban (1.táblázat; S3 táblázat). Az X es Y kromoszómák lefedettségének kiszámítása a kromoszóma teljes hosszának figyelembe vételével történt. Az Y és X kromoszómákra illeszthető szekvenciák arányából minden esetben egyértelműen meghatároztuk az egyének kromoszómális nemét. A mitokondriális genom átlagos lefedettsége minden mintánál legalább 100-szoros volt. Kivételt jelentett ez alól a HUAA (Antiochiai Anna) minta, melynél csak 23-szoros átlagos lefedettséget értünk el. Az ANGSD X módszer alapján a becsült X kromoszóma szennyeződés mértéke a mintákban 0,3% és 1,34% között változott, kivéve a HU53 minta 11,94%-os értékét. A mitokondriális szennyeződés Schmutzi programmal való vizsgálata során szintén a HU53 minta esetén tapasztaltunk 5%-os kiugró szennyezettséget a többi mintánál megfigyelt 1-2%-os értékhez képest (1. táblázat).
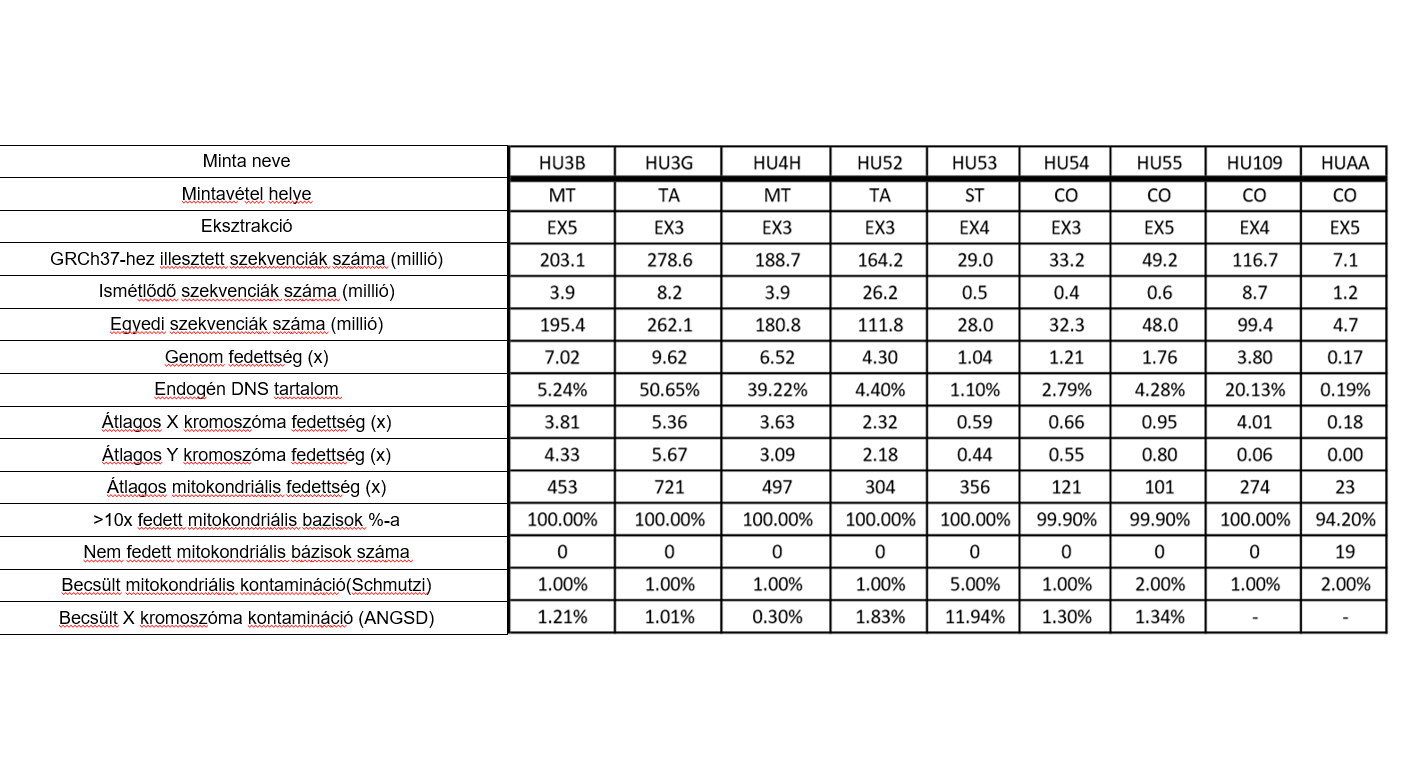
Az adatok mennyisége és minősége minden mintánál lehetővé tette az Y kromoszóma és a mitokondriális haplocsoport meghatározását.
Az archaikus minták Y kromoszóma haplocsoportjának meghatározása
A Yleaf szoftver ISOGG (International Society of Genetic Genealogy; Genetikai Származáskutatás Nemzetközi Társasága) formátumban adja meg a férfi minták Y kromoszómás haplocsoportját (2. táblázat és S4 táblázat). Az adatok Family Tree haplocsoportokon és SNP információkon alapuló manuális értékelésével azonban minden esetben a Yleaf által meghatározottaknál mélyebb besorolást értünk el. Ezért az egyes egyének haplocsoport meghatározásánál az ISOGG alapú haplocsoporthoz mellékeltük a Family Tree alapú mélyebb besorolás eredményét is (2. ábra, 2. táblázat és S5 táblázat).
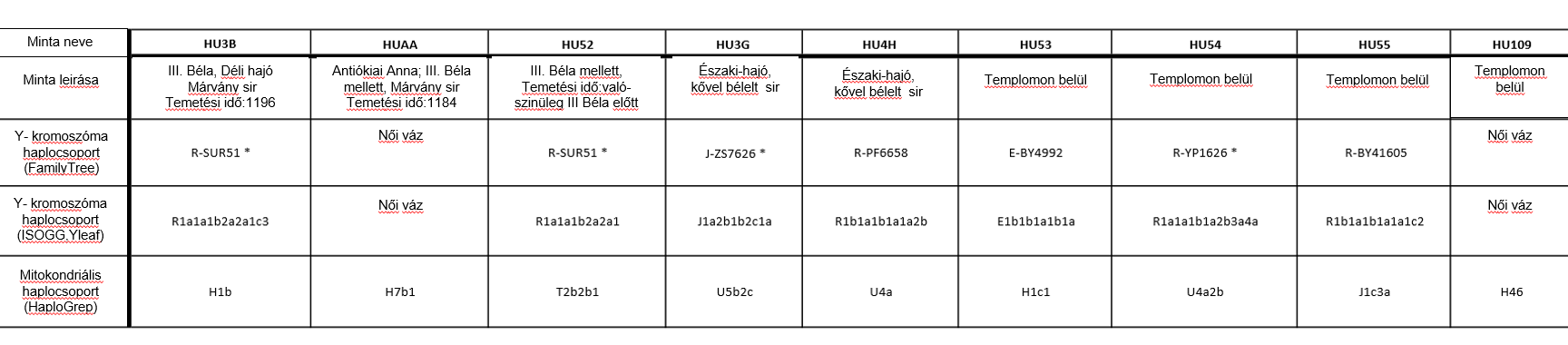
A HU53 mintát a BY4991 és BY4999 elágazási pont SNP-ek alapján az E-BY4992 (E1b1b1a1b1a) haplocsoportba soroltuk. Ez a haplocsoport gyakori a Dél- Balkánon, különösen Görögországban (32), de azonosították honfoglaló magyarokban és avarokban egyaránt (6).
A HU3G mintát a J-ZS7626 (J1a2b1b2c1a2b) haplocsoportba soroltuk, mivel a 25 haplocsoport definiáló SNP-ből 6 SNP-t azonosítottunk benne. Ez a haplocsoport Kelet-Afrikától az Arab-félsziget déli részéig elterjedt, de a Kaszpi-tenger nyugati partján élő tabasaran, kalmük, avar és lezgin populációkban is előfordul (14,33).
A vizsgált maradványok között leggyakoribb haplocsoport az eurázsiai elterjedésű R1 haplocsoport, amely nyugati (R1b) és keleti (R1a) ágra bontható (34,35).
A HU55 minta az R-BY41605 (R1b1a1b1a1a1c2) haplocsoportba tartozik. Az R-BY41605 haplocsoportot megelőző R-U106 haplocsoport Nyugat-Európában a leggyakoribb (34), de viszonylag gyakori a honfoglaló magyarok között is (6).
A HU4H minta az R-PF6658 (R1b1a1b1a1a2b) haplocsoportba sorolható. Habár az egyén feltehetően az R-BY3642 haplocsoportba tartozik, ennek az SNP-nek a leolvasási minősége nem teszi lehetővé az egyértelmű besorolást. Az R-PF6658 haplocsoportból származtatható R-BY3636, R-BY3630, R-BY3851, R-FGC30121, R-BY42688 és R-Y16335 haplocsoport meghatározó SNP-k nem voltak jelen, így ezeket az alágakat kizárhatjuk. Az R1b-U152, amely az R-PF6658-t megelőző haplocsoport, főként Olaszország alpesi vidékeire és Korzikára korlátozódik (34).
A HU3B, HU52 és HU54 minták az R1a haplocsoportba tartoznak. A HU54 az R-YP1626 (R1a1a1b1a2b3a4a2c) haplocsoportba tartozik, amely legáltalánosabban Délnyugat-Oroszországban, Ukrajnában, Fehéroroszországban és Kelet-Lengyelországban fordul elő (35).
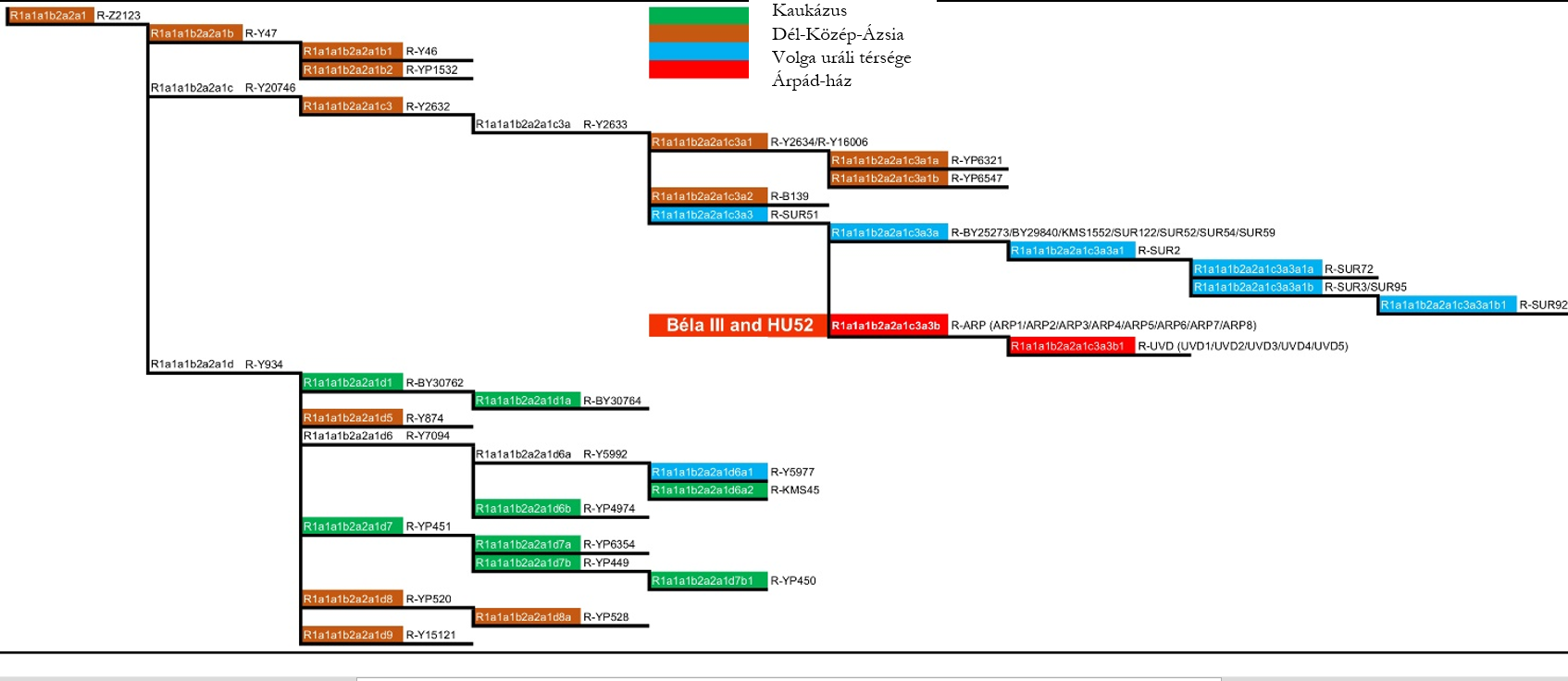
III. Béla (HU3B) és a HU52 egyén maradványai egyaránt az R-Z2125 (R1a1a1b2a2a) haplocsoportba tartoznak. Ez a haplocsoport általános Északkelet-Afganisztánban, Tádzsikisztánban, Kirgizisztánban és Dél-Kazahsztánban, valamint ritkábban előfordul a Volga-Urál régióban, a Kaukázusban és Iránban (1. ábra) (35). Az Árpád-ház apai vonalának meghatározását tovább pontosítottuk az R-Y2632, R-Y20746, R-Y2633 haplocsoportokat meghatározó SNP-ékkel, és az R-SUR51 haplocsoportot meghatározó SNP-ek közül 16-tal (2. ábra, 3. ábra, S5 táblázat) (13,14,16,36,37).
Az R-Z2123 haplocsoport nagy felbontású elemzése
A Volga-Urál régióból, a Kaukázus vidékéről és Közép-Ázsiából, 40 populációból összesen 4340 mintát vizsgáltunk meg az R-Z2125 és R-Z2123 markerekre (1. ábra, S1 táblázat). Több mint 400 mintát az R-Z2125 haplocsoportba, es ezen belül 320 mintát az R-Z2123 haplocsoportba soroltunk. 206 reprezentatív R-Z2123 mintát választottunk ki teljes genom szekvenálásra, hogy biztosítsuk a haplocsoport nagy felbontású besorolását (2. és 3. ábra). Vizsgálatainkba bevontunk két korábban már publikált Y kromoszóma szekvenciát (egy iraki és egy iraki zsidó egyén mintái) valamint a két árpád-házi egyén, III. Béla és HU52 mintáit (16) (S6 táblázat). A 210 vizsgált mintából 5 afgán és egy csecsen egyént nem sikerült az R-Z2123* (R1a1a1b2a2a1) haplocsoporton túl meghatározni (nem tartalmazták az ismert ágakra jellemző SNP-eket). 12 afganisztáni minta az R-Y47 (R1a1a1b2a2a1b) haplocsoportba tartozott. 58 egyént, közöttük 48, zömében a Burzjan és Abzelil régióból származó baskírt, 4 afgánt, a két árpád-házi személyt, egy szerbiai egyént, egy Lahore-i punjabit (HG03636)(13), egy iraki zsidó (GRC14414377 a.k.a 16198) (14, 16, 36) és egy iraki mintát (GRC15570738) (16) az R-Y2632 (R1a1a1b2a2a1c3) haplocsoportba soroltunk. Míg az iraki egyént nem tudtuk mélyebben besorolni, addig a másik 57 minta az R-Y2633 (R1a1a1b2a2a1c3a) haplocsoportba tartozott. Ez követően a minták három haplocsoportra oszlottak: a B139 SNP-t kizárólag az iraki zsidó egyén hordozta; az Y2634 és Y16006 markerek 4 afgán, valamint egy punjabi mintában voltak azonosíthatók; a SUR51 markerek 48 baskír, egy szerbiai és a két árpád-házi egyén mintájában voltak jelen. Az R-SUR51 haplocsoport 17 közös SNP-t követően két ágra bomlik (S7 táblázat). Az egyik ágán a baskír minták találhatóak, melyek Y kromoszómája további 6 SNP-t tartalmaz, amelyeket eddig a SUR51 haplocsoportba soroltak, míg a szerb egyén és a két árpád-házi személy szekvenciájából hiányzik ez a 6 SNP, azonban hordoznak 8 másik variaciót, amelyek az R-ARP haplocsoportot határozzák meg. A szerbiai egyén további 9 SNP-pel rendelkezik, amelyek hiányoznak az árpád-házi mintákból, és igy egy új haplocsoportot, az R-UVD-t (Újvidék; Novi Sad magyar neve) határozzák meg. 38 baskír egyén az R-SUR2 haplocsoportba és ennek al-haplocsoportjaiba az R-SUR72-be és az SUR95/R-SUR3-be tartozik, míg 7 egyént nem tudtunk az R-SUR51* haplocsoportnál mélyebben besorolni. A másik 134 egyén, köztük 15 afgán minta, 89 kaukázusi, harminc, többnyire az Arhangelski régióból származó baskír, az R-Y934 haplocsoportba és al-haplocsoportjaiba tartoznak (2. és 3. ábra, S6 táblázat).
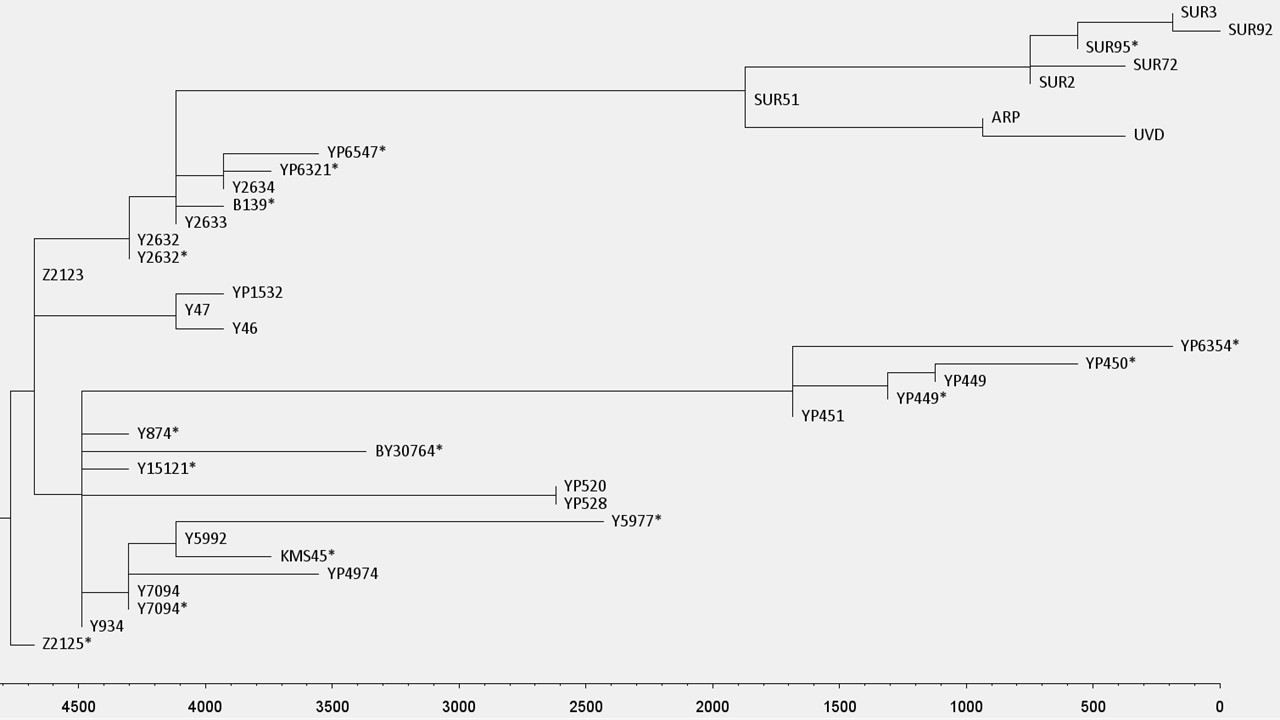
A Clustal Omega (v.1.2.4) szoftver segítségével filogenetikai fát építettünk a 208 jelenkori minta és a két árpád-házi személy szekvenciái alapján, hogy meghatározzuk a leszármazási viszonyaikat és az utolsó közös ős becsült korát (3. ábra). Az időskála meghatározásához az R-Y2633 haplocsoport 4100 évvel ezelőtti megjelenését vettük alapul (16). Az adatok alapján a szerbiai egyén és az árpád-házi személyek ága körülbelül 2000 éve vált el a baskírokétól, míg az R-Z2123 haplocsoport négy, e vizsgált populációban reprezentált ága, az R-Z2123*, R-Y47, R-Y2632 és R-Y934 haplocsoport, hozzávetőleg 4500 évvel ezelőtt jelentek meg.
Diszkusszió
Célunk az Árpád-házat meghatározó SNP-ek azonosítása, valamint az Árpád-ház filogenetikai eredetének meghatározása volt. Az Olasz és mtsai. által korábban Y-STR markerekre vizsgált hét ősi férfi csontváz Y-kromoszóma SNP markeren alapuló haplocsoport elemzése megerősíti hogy III. Bélán kívül csak a HU52 megjelölésű minta tartozik az Árpád-házhoz, mivel csak ez a minta ossza az R-ARP haplocsoportot III. Béla királlyal (7). A többi egyed különböző származású (J-ZS7626, E-BY4992, R286 BY41605, R-BY3642 and R-Y2608) és nem álltak rokonságban apai ágon sem a királyokkal, sem egymással. A mitokondriális haplocsoport-elemzés egymástól különböző haplocsoportokat tárt fel az összes minta esetében, jelezve, hogy nincs köztük anyai ági rokonság (2. táblázat és S2 táblázat). Az a tény, hogy sem Antióchiai Anna, sem pedig III. Béla nem rendelkezik a HU52-vel azonos mitokondriális haplocsoporttal, kizárja annak lehetőségét, hogy HU52 a fiuk vagy III. Béla fiútestvére lenne. HU52 feltehetően III. Béla apja, II. Géza király (1130–1162), vagy nagyapja, II. Béla (1110–1141) vagy egy még korábbi ős.
Az Árpád-ház két tagjának azonosítása lehetővé tette, hogy megkíséreljük a dinasztia filogenetikai eredetének meghatározását. 17 közös R-SUR51 SNP alapján megállapítottuk, hogy a vizsgált népcsoportok közül a baskírok, elsősorban a Burzjan és Abzelil járásokból, az Árpád-ház legközelebbi rokonai (S7 táblázat). Ezután továbbszármaztattuk az Árpád-ház Y-kromoszómás haplocsoportját a köztük és egy a mai Szerbiából származó egyén 9 közös SNP-je alapján. Ezt a haplocsoportot R-ARP-nak neveztük el (ÁRPád -haplocsoport; R1a1a1b2a2a1c3a3b az ISOGG névadási konvenciót követve).
Számos szerző megegyezik abban, hogy egy új Y-kromoszóma SNP 100-150 évenként stabilizálódik a csírasejtvonalban (16). E kalibráció alapján Behar és mtsai. (2017) a fent említett iraki és az iraki zsidó vonalak elválásának, azaz a Y2633 SNP megjelenésének idejét körülbelül 4100 évvel ezelőttre teszik (16). Ha ezt a dátumot kiindulási pontként kezeljük, a Clustal Omega szoftver felhasználásával készült koaleszcens idő becslés arra utal, hogy a Z2123* haplocsoport robbanás szérű szétágazása (az R-YP3920, R-YP4907, R-Y47, R-Y934, R-Y2632 haplocsoportok megjelenése) a jelentől számítva legalább 4500 évvel ezelőtt történt (3. ábra). Mivel az adathalmazunkból származó összes Z2123* egyed és a tovább deriválható korai ágakhoz, az R-Y47-hez (R-46), az R-Y934-hez (R-Y874*, R-Y15121*, R-YP520*), és az R-Y2632-hez (R-Y2634 and R-Y16006; R-YP6321*; R-YP6547*) tartozó minták többsége Afganisztán területéről származik (25), igy ez az esemény valószínűleg egy észak-Afganisztán központú régióban történt. Az R-Y2632 és R-Y934 haplocsoportok modernebb ágai, az R-SUR51, illetve az R-YP451 és R-Y5977 haplocsoportok, a Volga uráli szakaszán, illetve a kaukázusi régióban találhatók. Ez egy újabb keletű népességbővülést sugall ezekben a jelenkori régiókban.
Elemzésünk kimutatja, hogy a modernkori baskírok leszármazási vonala körülbelül 2000 évvel ezelőtt vált el az Árpádok vonalától ahogy ezt az R-ARP haplocsoport megjelenése mutatja. A mai Szerbiából származó egyedben jelenlévő R-UVD haplocsoport megjelenése becslésünk szerint 900 évvel ezelőtt történt, mely időpont megfelel III. Béla király temetkezési dátumának (1196). Természetesen ezek a számok csak becslések, és nagymértékben azoktól az SNP-knek a számától függenek, amelyeket jelenleg az ilyen típusú számításokban felhasználhatóknak és megbízhatóknak tartanak (S8 táblázat). Az általunk beazonosított, összesen 9 R-ARP SNP közül csak 8 esik az ajánlott régióba, míg a 9 UVD SNP közül csak 6-ot tudtunk felhasználni az elemzésünkben. Véleményünk szerint a jobb Y-kromoszóma referencia szekvenciák alapján felül kellene vizsgálni az Y-kromoszóma régiót, ahonnan SNP-ket figyelembe lehet és kell venni a filogenetikai fák felállításához.
A Kárpát-medencét elfoglaló magyarok filogenetikai származása vitatott (40). Nyelvészeti érvek alapján az a hipotézis került előtérbe, hogy elsősorban egy finnugor nyelvet beszélő népességet képviseltek, miközben az Árpád-ház szóbeli és írásos hagyománya a hunokkal való rokonságra utal. Az Árpád-ház két tagjának genetikai elemzése alapján úgy tűnik, hogy olyan filogenetikai vonalból származtak (RZ2125), amely ma elsősorban iráni vagy török nyelvű etnikai csoportok körében van jelen (pástú, tádzsik, türkmén, üzbég, baskír). Mindazonáltal a legközelebbi rokonok, a baskírok közel élnek finnugor nyelvet beszélő népekhez, amelyek között gyakori az N-B539 haplocsoport. Egy közelmúltban végzett kutatás kimutatta, hogy ez a haplocsoport jelenkori magyarokban is jelen van (41). Érdekes módon a jelenkori magyarokban és baskírokban talált N-B539-es haplocsoport utolsó szétválása a becslések szerint körülbelül 2000 évvel ezelőtt történt (42). Ez arra utal, hogy egy részben török (R-SUR51) és részben finnugor (N-B539) komponensből álló embercsoport körülbelül 2000 évvel ezelőtt hagyta el a Volga uráli régióját és kezdte el vándorlását, amely végül a Kárpát-medencei letelepedéshez vezetett. Mind a jelenlegi Kárpát-medencei népesség körében, mind a magyar honfoglalás időszakából származó temetkezési maradványok esetében nagyobb felbontású N-B539, R-SUR51 és R-ARP haplocsoport analízisre van szükség ahhoz, hogy ezt a feltételezést igazolni tudjuk. Célzott mintavétel a Fekete-tenger és a Don (vagy Dnyeper) folyó és a Keleti-Kárpátok közötti területről, az ősi Levédia és Etelköz térségeiből, további adatokat szolgáltathatnak a Kárpát-medencei letelepedés előtti magyar népvándorlások feltérképezésére. Az Árpád-ház Y-kromoszóma vonalának ritka mivolta lehetővé teszi majd a magyar őstörténet igen részletes és pontos feltérképezését, valamint a dinasztia további leszármazottainak azonosítását, ami a téma iránt érdeklődő tudósok fő célkitűzése volt évszázadokon át.
Köszönetnyilvánítás
A szerzők szeretnék kifejezni hálás köszönetüket Erdő Péter bíborosnak, Esztergom-Budapest érsekének, hogy engedélyezte az emberi maradványok exhumálását genetikai elemzés céljából, valamint köszönetünket fejezzük ki Dr. Török Tibornak a kézirat szakmai lektorálásáért.
Érdekellentét:
A szerzők és társszerzők ezúttal nyilatkoznak arról, hogy nem áll fenn érdekellentét.
Finanszírozás:
Az NGS szekvenálást és adatelemzést az Emberi Erőforrások Minisztériuma finanszírozta (EMMI) a 26090/2019/MINKABINET számú projekt keretében. Neparáczki Endre a következő támogatásokban részesült: 62722/2018 számú tanúsítvány és a 361/2019 (TUDFO/5157-1/2019-ITM) kiválósági program.
Doron Behart az Észt Kutatási Tanács IUT-24 részleges támogatását élvezte.
Elza Khusnutdinova és Natalia Ekomasova a következő, az Orosz Kutatási Alapítvány által biztosított támogatásokban részesült: 17-44-020748 és 19-364 04-01195
Murat Dzhaubermezov az Orosz Tudományos es Felsőoktatási Miniszterium Kutatási Alapítványa által biztosított támogatásban részesült: FZWU-2020-0027
Julie di Cristofaro és Jacques Chiaroni munkásságát részben az Agence National de la 366 Recherche (Grant #BLAN07-3_222301, CSD 9 – Sciences humaines et sociales) támogatta.
A támogatók nem befolyásolták a kutatás projekt kialakítását, az adatok gyűjtését és elemzését, a publikációs szándékot vagy a kézirat elkészültét.
Kiegészítő információ:
A kiegészítő információk és táblázatok EJHG honlapján találhatók.
Adatok elérhetősége:
A tanulmányban bemutatott összes minta mitokondriális és Y-kromoszómás szekvenciáinak BAM fájljai elérhetők a következő linken: https://www.ncbi.nlm.nih.gov/bioproject/PRJNA490697: Determination of the phylogenetic
379 origins of the Árpád Dynasty based on Y chromosome sequencing of Béla the Third
Kiegészítő információ
S1. ábra. A székesfehérvári bazilikából származó 9 felnőtt csontváz mitokondriális haplocsoport meghatározása
S2 ábra. A székesfehérvári bazilikából származó 7 felnőtt férfi csontváz Y-kromoszóma haplocsoport meghatározása
S1 táblázat: 54 különböző begyűjtési helyszínről származó, 40 különböző népességet képviselő 4340 jelenkori minta haplocsoport besorolása
S2 táblázat: A HaploGrep (24) által használt mitokondriális SNP-ek listája az rCRS referencia alapján (van Oven M, Kayser M. 2009 ; 44).
S3 táblázat: Archaikus minták NGS statisztikai jellemzői a GRCh37 referenciához való illesztés alapján.
S4 táblázat: Archaikus minták Y kromoszóma haplocsoport meghatározása a Yleaf program segítségével.
S5 táblázat: Az archaikus minták haplocsoportjának meghatározásában használt Family Tree SNP-ek listája, a szekvencia minőségi mutatóival.
S6 táblázat: Jelenkori minták NGS statisztikai jellemzői és a legmagasabb felbontása haplocsoport analízise a Family Tree haplocsoport névhasználata alapján
S7 táblázat: Az ARP és UVD haplocsoportok definiálása.
S8 táblázat: A Z2123 haplocsoport fájának elkészítésében használt SNP-ek listája
Irodalomjegyzék
1. Hóman B. Geschichte des Ungarischen Mittelalters. Berlin: Walter de Gruyter; 1940-43.
2. Neparaczki E, Juhasz Z, Pamjav H, Feher T, Csanyi B, Zink A, et al. Genetic structure of the early Hungarian conquerors inferred from mtDNA haplotypes and Y-chromosome haplogroups in a small cemetery. Mol Genet Genomics. 2017;292(1):201-14.
3. Neparaczki E, Kocsy K, Toth GE, Maroti Z, Kalmar T, Bihari P, et al. Revising mtDNA haplotypes of the ancient Hungarian conquerors with next generation sequencing. PLoS One. 2017;12(4):e0174886.
4. Tomory G, Csanyi B, Bogacsi-Szabo E, Kalmar T, Czibula A, Csosz A, et al. Comparison of maternal lineage and biogeographic analyses of ancient and modern Hungarian populations. Am J Phys Anthropol. 2007;134(3):354-68.
5. Csosz A, Szecsenyi-Nagy A, Csakyova V, Lango P, Bodis V, Kohler K, et al. Maternal Genetic Ancestry and Legacy of 10(th) Century AD Hungarians. Sci Rep. 2016;6:33446.
6. Neparaczki E, Maroti Z, Kalmar T, Maar K, Nagy I, Latinovics D, et al. Y-chromosome haplogroups from Hun, Avar and conquering Hungarian period nomadic people of the Carpathian Basin. Sci Rep. 2019;9(1):16569.
7. Olasz J, Seidenberg V, Hummel S, Szentirmay Z, Szabados G, Melegh B, et al. DNA profiling of Hungarian King Béla III and other skeletal remains originating from the Royal Basilica of Székesfehérvár. Archaeological and Anthropological Sciences. 2019;11(4):1345-57.
8. Szentpétery I. Scriptores Rerum Hungaricarum. Budapest: Academia Litteraria Hungarica; 1937-38.
9. Engel P. Temetkezések a középkori székesfehérvári bazilikában [Burials in the medieval Basilica of Székesfehérvár]. Századok. 1987;121:613-37.
10. Érdy J. III. Béla király és nejének Székes-Fehérvárott talált síremlékei [The tombs of king Béla III and his spouse found in Székes-Fehérvár]. Pest: Emich G; 1853.
11. A székesfehérvári királyi bazilika embertani leletei 1848-2002: Balassi Kiadó, Budapest; 2008.
12. Poznik GD, Henn BM, Yee MC, Sliwerska E, Euskirchen GM, Lin AA, et al. Sequencing Y chromosomes resolves discrepancy in time to common ancestor of males versus females. Science. 2013;341(6145):562-5.
13. Poznik GD, Xue Y, Mendez FL, Willems TF, Massaia A, Wilson Sayres MA, et al. Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences. Nat Genet. 2016;48(6):593-9.
14. Karmin M, Saag L, Vicente M, Wilson Sayres MA, Jarve M, Talas UG, et al. A recent bottleneck of Y chromosome diversity coincides with a global change in culture. Genome Res. 2015;25(4):459-66.
15. International Society of Genetic Genealogy. Y-DNA Haplogroup Tree 2019, Version:14.22, Date: 25 January 2019, http://www.isogg.org/tree/
16. Behar DM, Saag L, Karmin M, Gover MG, Wexler JD, Sanchez LF, et al. The genetic variation in the R1a clade among the Ashkenazi Levites' Y chromosome. Sci Rep. 2017;7(1):14969.
17. Inc. I. Basespace application [Available from: www.illumina.com/BaseSpaceApps.
18. Bioinformatics B. Trim Galore [Available from: http://www.bioinformatics.babraham.ac.uk/projects/trim_galore/)
19. Miller NA, Farrow EG, Gibson M, Willig LK, Twist G, Yoo B, et al. A 26-hour system of highly sensitive whole genome sequencing for emergency management of genetic diseases. Genome Med. 2015;7:100.
20. Robinson JT, Thorvaldsdottir H, Winckler W, Guttman M, Lander ES, Getz G, et al. Integrative genomics viewer. Nat Biotechnol. 2011;29(1):24-6.
21. Jonsson H, Ginolhac A, Schubert M, Johnson PL, Orlando L. mapDamage2.0: fast approximate Bayesian estimates of ancient DNA damage parameters. Bioinformatics. 2013;29(13):1682-4.
22. Renaud G, Slon V, Duggan AT, Kelso J. Schmutzi: estimation of contamination and endogenous mitochondrial consensus calling for ancient DNA. Genome Biol. 2015;16:224.
23. Rasmussen M, Guo X, Wang Y, Lohmueller KE, Rasmussen S, Albrechtsen A, et al. An Aboriginal Australian genome reveals separate human dispersals into Asia. Science. 2011;334(6052):94-8.
24. Weissensteiner H, Pacher D, Kloss-Brandstatter A, Forer L, Specht G, Bandelt HJ, et al. HaploGrep 2: mitochondrial haplogroup classification in the era of high-throughput sequencing. Nucleic Acids Res. 2016;44(W1):W58-63.
25. Ralf A, Gonzalez DM, Zhong K, Kayser M. Yleaf: Software for Human Y-Chromosomal Haplogroup Inference from Next-Generation Sequencing Data. Mol Biol Evol. 2018;35(7):1820.
26. Y-DNA Haplogroup Tree 2019: International Society of Genetic Genealogy; 2020 [Available from: http://www.isogg.org/tree.
27. Y-chromosome DNA haplotree: Family Tree DNA Ltd.; [Available from: https://www.familytreedna.com/public/y-dna-haplotree.
28. Au CH, Ho DN, Kwong A, Chan TL, Ma ESK. BAMClipper: removing primers from alignments to minimize false-negative mutations in amplicon next-generation sequencing. Sci Rep. 2017;7(1):1567.
29. Suchard MA, Lemey P, Baele G, Ayres DL, Drummond AJ, Rambaut A. Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10. Virus Evol. 2018;4(1):vey016.
30. Sievers F, Wilm A, Dineen D, Gibson TJ, Karplus K, Li W, et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Mol Syst Biol. 2011;7:539.
31. FigTree v1.4.4.
32. Cruciani F, La Fratta R, Trombetta B, Santolamazza P, Sellitto D, Colomb EB, et al. Tracing past human male movements in northern/eastern Africa and western Eurasia: new clues from Y-chromosomal haplogroups E-M78 and J-M12. Mol Biol Evol. 2007;24(6):1300-11.
33. Balanovsky O, Dibirova K, Dybo A, Mudrak O, Frolova S, Pocheshkhova E, et al. Parallel evolution of genes and languages in the Caucasus region. Mol Biol Evol. 2011;28(10):2905-20.
34. Myres NM, Rootsi S, Lin AA, Jarve M, King RJ, Kutuev I, et al. A major Y-chromosome haplogroup R1b Holocene era founder effect in Central and Western Europe. Eur J Hum Genet. 2011;19(1):95-101.
35. Underhill PA, Poznik GD, Rootsi S, Jarve M, Lin AA, Wang J, et al. The phylogenetic and geographic structure of Y-chromosome haplogroup R1a. Eur J Hum Genet. 2015;23(1):124-31.
36. Rootsi S, Behar DM, Jarve M, Lin AA, Myres NM, Passarelli B, et al. Phylogenetic applications of whole Y-chromosome sequences and the Near Eastern origin of Ashkenazi Levites. Nat Commun. 2013;4:2928.
37. Muratov. The genus of Shagali Shakman, the clan of Olobure and the descendants of Inas (Kipchak Khan) according to Big-Y. Bulletin of the EI Project 'Suyun'. 2014;1(1).
38. Di Cristofaro J, Pennarun E, Mazieres S, Myres NM, Lin AA, Temori SA, et al. Afghan Hindu Kush: where Eurasian sub-continent gene flows converge. PLoS One. 2013;8(10):e76748.
39. Zgonjanin D, Alghafri R, Antov M, Stojiljkovic G, Petkovic S, Vukovic R, et al. Genetic characterization of 27 Y-STR loci with the Yfiler((R)) Plus kit in the population of Serbia. Forensic Sci Int Genet. 2017;31:e48-e9.
40. Neparaczki E, Maroti Z, Kalmar T, Kocsy K, Maar K, Bihari P, et al. Mitogenomic data indicate admixture components of Central-Inner Asian and Srubnaya origin in the conquering Hungarians. PLoS One. 2018;13(10):e0205920.
41. Post H, Nemeth E, Klima L, Flores R, Feher T, Turk A, et al. Y-chromosomal connection between Hungarians and geographically distant populations of the Ural Mountain region and West Siberia. Sci Rep. 2019;9(1):7786.
42. Amorim CEG, Vai S, Posth C, Modi A, Koncz I, Hakenbeck S, et al. Understanding 6th-century barbarian social organization and migration through paleogenomics. Nat Commun. 2018;9(1):3547.
43. Team QD. QGIS Geographic Information System; Open Source Geospatial Foundation Project. 2015 [Available from: http://www.qgis.org.
44. van Oven M, Kayser M. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation. Hum Mutat. 2009;30(2):E386-94.